Впр сравнение двух таблиц: Как пользоваться функцией ВПР в Excel: пример с двумя таблицами
Содержание
Сравнение 2 файлов в Excel на предмет различий
Автор Амина С. На чтение 10 мин Опубликовано
Каждый пользователь может столкнуться с ситуацией, когда нужно сравнить две таблицы. Ну в крайнем случае всем приходится сравнивать две колонки. Да, безусловно, работа с файлами Excel отличается высоким удобством и комфортом. Жаль, это сравнения не касается. Конечно, возможна визуальная сортировка таблицы небольшого размера, но когда количество ячеек идет на тысячи, то приходится пользоваться дополнительным аналитическим инструментарием.
К сожалению, еще не была открыта магическая палочка, позволяющая в один клик сравнить автоматически всю информацию между собой. Поэтому приходится поработать, а именно, собрать данные, указать необходимые формулы и выполнить другие действия, позволяющие хоть немного автоматизировать сравнения.
Таких действий множество. Давайте рассмотрим некоторые из них.
Давайте рассмотрим некоторые из них.
Содержание
- С какой целью проводится сравнение файлов Excel
- Все способы сравнения 2 таблиц в Excel
- Формула равенства и проверка на ложь-истину
- Выделение различающихся значений
- Сравнение 2 таблиц при помощи условного форматирования
- Функция СЧЁТЕСЛИ + правила для сравнения таблиц
- Функция ВПР для сравнения 2 таблиц
- Функция ЕСЛИ
- Макрос VBA
- Как провести сравнение файлов в Эксель
- Условное форматирование для сравнения 2 файлов Эксель
- Сравнение данных в Эксель на разных листах
- Как сравнить 2 листа в таблице Эксель
- Средство сравнения электронных таблиц
- Как интерпретировать результаты сравнения
С какой целью проводится сравнение файлов Excel
Причин, по которым осуществляется сравнение нескольких экселевских файлов, может быть огромное множество. Рано или поздно каждый пользователь сталкивается с такой необходимостью, и таких вопросов у него не возникает. Например, может понадобиться сравнить данные из двух отчетов за разные кварталы на предмет того, выросли финансовые показатели или уменьшились.
Например, может понадобиться сравнить данные из двух отчетов за разные кварталы на предмет того, выросли финансовые показатели или уменьшились.
Или, как вариант, преподавателю необходимо посмотреть, каких учеников выгнали из университета, сравнив состав студенческой группы в прошлом году и этом.
Таких ситуаций может быть огромное количество. Но давайте перейдем к практике, потому что тема довольно сложная.
Все способы сравнения 2 таблиц в Excel
Хотя тема сложная, но она легкая. Да, не стоит удивляться. Сложная она, потому что складывается из множества частей. Но сами эти части легкие для восприятия и выполнения. Давайте рассмотрим, как можно сравнивать две экселевские таблицы, непосредственно на практике.
Формула равенства и проверка на ложь-истину
Начнем, конечно, с наиболее простого метода. Таким методом сравнение документов возможно, причем в довольно широких пределах. Можно сопоставлять между собой не только текстовые значения, но и числовые.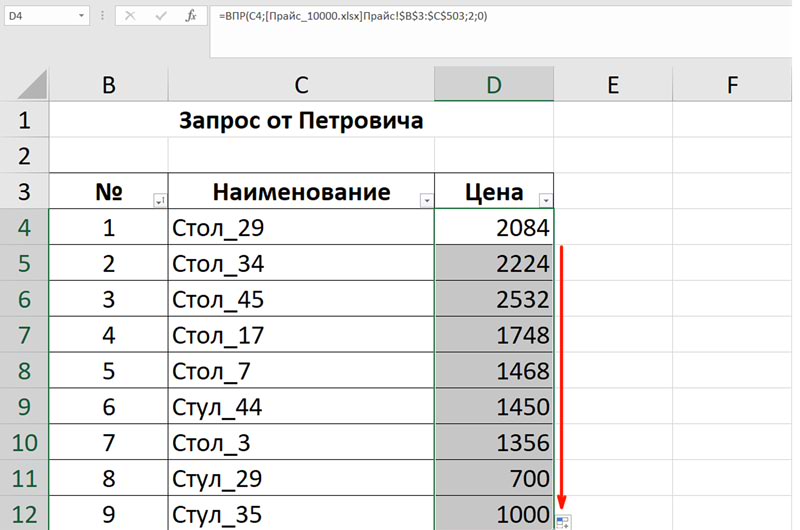 И давайте приведем небольшой пример. Допустим, у нас есть два диапазона с ячейками числового формата. Для этого достаточно просто написать формулу равенства =C2=E2. Если окажется, что они равны, в ячейке будет написано «ИСТИНА». Если же они будут отличаться, то «ЛОЖЬ». После этого нужно данную формулу перенести на весь диапазон с помощью маркера автозаполнения.
И давайте приведем небольшой пример. Допустим, у нас есть два диапазона с ячейками числового формата. Для этого достаточно просто написать формулу равенства =C2=E2. Если окажется, что они равны, в ячейке будет написано «ИСТИНА». Если же они будут отличаться, то «ЛОЖЬ». После этого нужно данную формулу перенести на весь диапазон с помощью маркера автозаполнения.
Теперь разница видна невооруженным глазом.
1
Выделение различающихся значений
Также можно сделать так, чтобы значения, которые отличаются между собой, были выделены особенным цветом. Это тоже довольно простая задача. Если вам достаточно найти отличия между двумя диапазонами значений или целыми таблицами, необходимо перейти на вкладку «Главная», и там выбрать пункт «Найти и выделить». Перед тем, как вы ее нажмете, не забудьте выделить набор ячеек, в которых сохраняется информация для сравнения.
В появившемся меню необходимо кликнуть по меню «Выделить группу ячеек…». Далее откроется окошко, в котором нам необходимо в качестве критерия выбрать отличия по строкам.
23
Сравнение 2 таблиц при помощи условного форматирования
Условное форматирование является очень удобным и, что важно, функциональным методом, позволяющим выбрать цвет, которым будет выделяться отличающееся или такое же значение. Найти этот параметр можно на вкладке «Главная». Там можно найти кнопку с соответствующим названием и в появившемся перечне выбираем «Управление правилами». Появится диспетчер правил, в котором нам надо выбрать меню «Создать правило».
4
Далее из перечня критериев нам нужно выбрать тот, где написано, что нужно использовать формулу для определения ячеек, которые будут отформатированы особенным образом. В описании правила нужно задать формулу. В нашем случае это =$C2<>$E2, после чего подтверждаем свои действия нажатием кнопки «Формат». После этого задаем внешний вид ячейки и смотрим, нравится ли он, через специальное мини-окошко с образцом.
Если все устраивает, нажимаем кнопку «ОК» и подтверждаем действия.
5
В диспетчере правил условного форматирования пользователь может найти все правила форматирования, действующие в этом документе.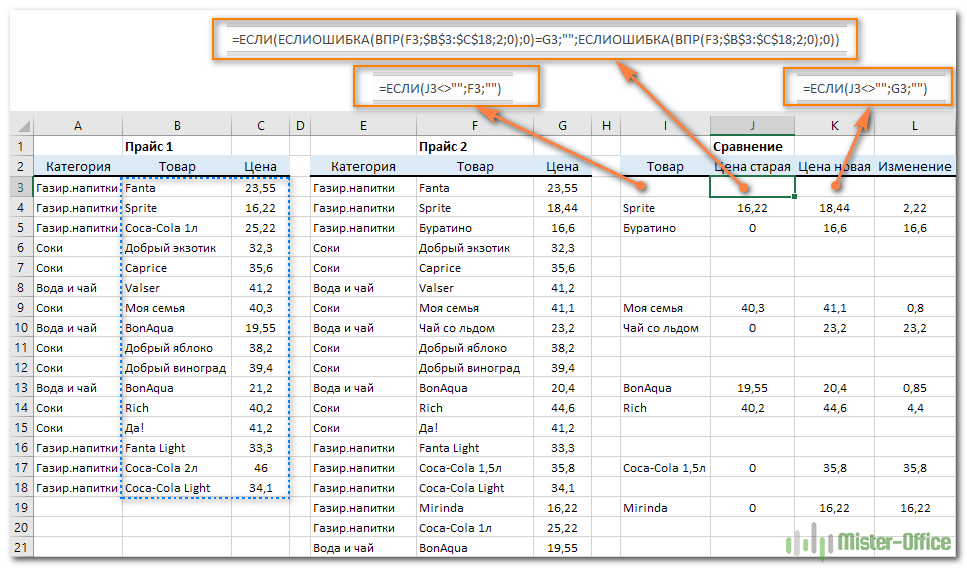
Функция СЧЁТЕСЛИ + правила для сравнения таблиц
Все методы, которые мы описали ранее, удобны для тех формат, формат которых един. Если же предварительно таблицы не были упорядоченными, то лучше всего метод, в котором необходимо сравнивать две таблицы с помощью функции СЧЕТЕСЛИ и правил.
Давайте вообразим, что у нас есть два диапазона с немного различающейся информацией. Перед нами стоит задача сравнивать их и понять, какое значение отличается. Для начала необходимо осуществить его выделение в первом диапазоне и перейти на вкладку «Главная». Там находим уже знакомый нам ранее пункт «Условное форматирование». Создаем правило и в качестве правила задаем использовать формулу.
В этом примере формула такая, как показана на этом скриншоте.
6
После этого задаем формат описанным выше способом. Эта функция анализирует значение, содержащееся в ячейке C1 и смотрит на диапазон, указанный в формуле. Он соответствует второму столбцу. Нам необходимо взять это правило, и скопировать на весь диапазон. Ура, все ячейки с неповторяющимися значениями выделяются.
Ура, все ячейки с неповторяющимися значениями выделяются.
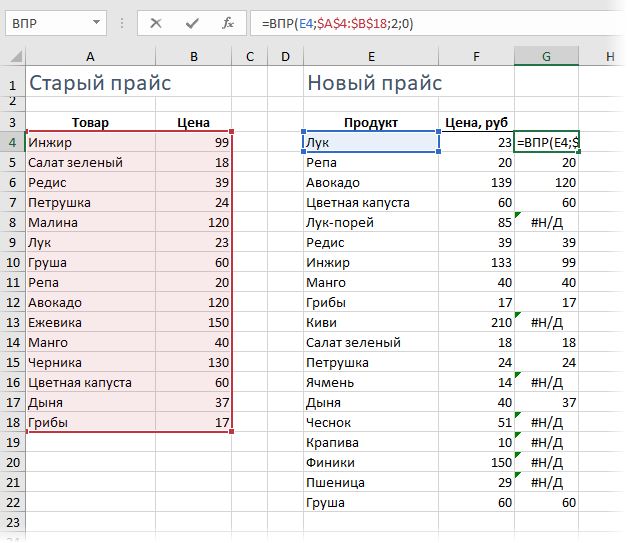
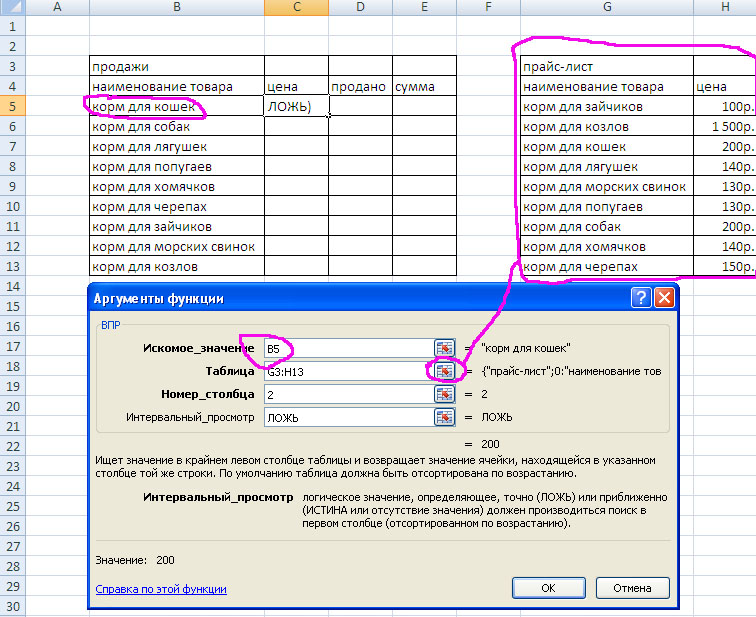
Функция ВПР для сравнения 2 таблиц
В данном методе нами будет рассматриваться функция ВПР, которая проверяет, нет ли совпадений в двух таблицах. Чтобы это сделать, необходимо ввести формулу, указанную на картинке ниже и перенести ее на весь диапазон, который используется для сравнения.
Эта функция перебирает каждое значение и смотрит, нет ли повторов из первой колонки во второй. Ну и после выполнения всех операций записывается в ячейке это значение. Если его там нет, то получаем ошибку #Н/Д, чего вполне достаточно для того, чтобы автоматически понимать, какое значение будет не соответствовать.
7
Функция ЕСЛИ
Логическая функция ЕСЛИ – это еще один неплохой способ сравнения двух диапазонов. Главная особенность этого метода заключается в том, что можно использовать лишь ту часть массива, которая сравнивается, а не вся таблица. Это экономит ресурсы как компьютера, так и пользователя.
Давайте приведем небольшой пример.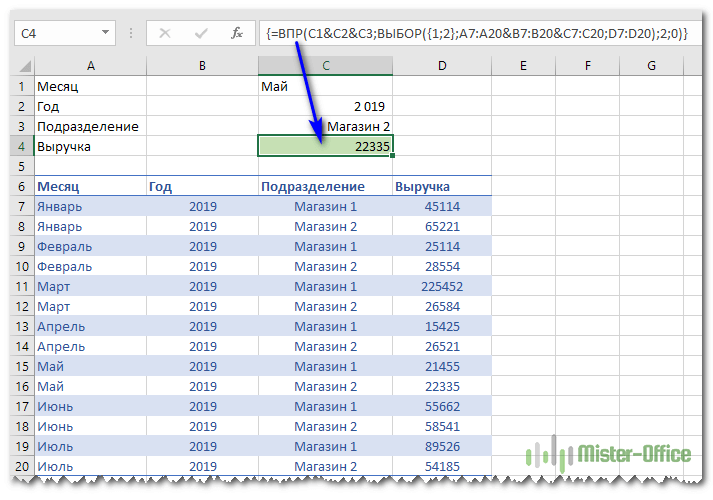 У нас есть два столбца – A и B. Нам нужно сравнить часть информации в них между собой. Для этого нам нужно подготовить еще один сервисный столбец C, в котором записывается следующая формула.
У нас есть два столбца – A и B. Нам нужно сравнить часть информации в них между собой. Для этого нам нужно подготовить еще один сервисный столбец C, в котором записывается следующая формула.
8
С помощью формулы, которая использует функции ЕСЛИ, ЕСЛИОШИБКА и ПОИСКПОЗ можно перебрать все нужные элементы колонки А, а потом в колонке B. Если оно было обнаружено в колонке B и A, то оно возвращается в соответствующую ячейку.
Макрос VBA
Макрос – это самый сложный, но и наиболее продвинутый метод сравнения двух таблиц. некоторые варианты сравнения вообще невозможны без сценариев VBA. Они позволяют автоматизировать процесс и сэкономить время. Все нужные операции для подготовки данных, если их запрограммировать один раз, будут выполняться и далее.
Основываясь на задаче, которую нужно решить, можно составлять любые программы, сравнивающие данные абсолютно без вмешательства пользователя.
Как провести сравнение файлов в Эксель
Если пользователь поставил перед собой задачу (ну или ему поставили ее) сравнить два файла, то это можно сделать сразу двумя методами. Первый из них – использование специализированной функции. Чтобы реализовать этот метод, следуйте инструкции:
Первый из них – использование специализированной функции. Чтобы реализовать этот метод, следуйте инструкции:
- Откройте те файлы, сравнение которых требуется.
- Откройте вкладку «Вид» – «Окно» – «Вид рядом».
После этого два файла будут открыты одном документе Excel.
Это же можно сделать банальными средствами Windows. Для начала необходимо открыть два файла в разных окнах. После этого взять одно окно и перетащить его в самую левую часть экрана. После этого открыть второе окно и перетащить его в самую правую часть. После этого два окна станут рядом.
Условное форматирование для сравнения 2 файлов Эксель
Очень часто сравнение документов подразумевает их выведение рядом друг с другом. Но в некоторых случаях возможна автоматизация этого процесса с помощью условного форматирования. С его помощью можно проверить, есть ли отличия между листами. Это позволяет выиграть время, которое можно использовать для других целей.
Сперва нам необходимо перенести сравниваемые листы в один документ.
Чтобы это сделать, необходимо нажать по подходящему листу правой кнопкой мыши, после чего нажать во всплывающем меню на кнопку «Переместить или скопировать». Далее появится диалоговое окно, в котором пользователь может выбрать документ, в который данный лист необходимо вставить.
9
Далее необходимо выделить все нужные ячейки, чтобы отобразить все различия. Проще всего это сделать, нажав самую верхнюю левую ячейку, после чего нажать на комбинацию клавиш Ctrl + Shift + End.
После этого переходим в окно условного форматирования и создаем новое правило. В качестве критерия используем подходящую в конкретном случае формулу, потом задаем формат.
Внимание: адреса ячеек нужно указывать те, которые на другом листе. Это можно сделать через меню ввода формулы.
Сравнение данных в Эксель на разных листах
Предположим, у нас есть перечень сотрудников, в котором также приводятся их заработные платы. Этот перечень обновляется каждый месяц. Этот список копируется на новый лист.
Этот список копируется на новый лист.
Предположим, нам нужно сравнить зарплаты. В таком случае можно в качестве данных использовать таблицы с разных листов. Для выделения различий будем использовать условное форматирование. Все просто.
С помощью условного форматирования можно осуществлять эффективное сравнение даже если фамилии работников располагаются в различном порядке.
Как сравнить 2 листа в таблице Эксель
Сравнение информации, располагающейся на двух листах, осуществляется с помощью функции ПОИСКПОЗ. В качестве первого ее параметра располагается пара значений, которую нужно искать на том листе, который отвечает за следующий месяц. Простыми словами, март. Мы можем обозначить просматриваемый диапазон, как совокупность ячеек, которые входят в состав именных диапазонов, объединенных попарно.
Так можно осуществить сравнение строк по двум критериям – фамилия и заработная плата. Ну или любым другим, определенным пользователем. Для всех совпадений, которые удалось найти, записывается в ячейке, в которую вводится формула, число.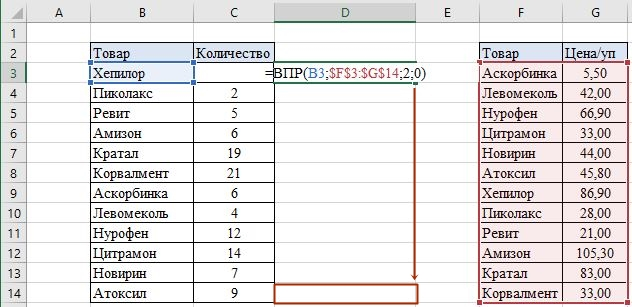 Для Excel такое значение всегда будет истинным. Поэтому для того, чтобы форматирование применялось по отношению к тем ячейкам, которые были различны, необходимо это значение заменить на ЛОЖЬ, воспользовавшись функцией =НЕ().
Для Excel такое значение всегда будет истинным. Поэтому для того, чтобы форматирование применялось по отношению к тем ячейкам, которые были различны, необходимо это значение заменить на ЛОЖЬ, воспользовавшись функцией =НЕ().
Средство сравнения электронных таблиц
В Excel заложен специальный инструмент, позволяющий сравнивать электронные таблицы и выделять изменения автоматически.
Важно учесть, что это средство доступно лишь для тех пользователей, кто купил офисные пакеты «Профессиональный плюс».
Открыть его можно непосредственно с вкладки «Главная», выбрав пункт «Сравнить файлы».
После этого появится диалоговое окно, в в котором нужно выбрать вторую версию книги. Также можно ввести адрес в интернете, по которому эта книга расположена.
После того, как мы выберем две версии документа, нужно подтвердить свои действия с помощью клавиши ОК.
В ряде случаев может быть выдана ошибка. Если она появляется, это может указывать на защищенность файла паролем. После того, как вы нажмете кнопку ОК, будет предложено указать его.
После того, как вы нажмете кнопку ОК, будет предложено указать его.
Средство сравнение выглядит, как две таблицы Excel, расположенные рядом друг с другом в рамках одного окна. В зависимости от того, информация была добавлена, удалена или было изменение формулы (а также других видов действий), изменения выделяются различными цветами.
Как интерпретировать результаты сравнения
Все очень просто: разные виды отличий обозначаются разными цветами. Форматирование может распространяться как на заливку ячейки, так и сам текст. Так, если в ячейку данные вводились, то заливка зеленого цвета. Если что-то становится непонятно, в самом сервисе есть обозначения, показывающие то, какой тип изменений каким цветом выделяется.
Оцените качество статьи. Нам важно ваше мнение:
Сравнение двух таблиц в Excel
Готовые решения »
Надстройка VBA-Excel »
Сравнение таблиц
Довольно часто приходится сравнивать таблицы Excel между собой.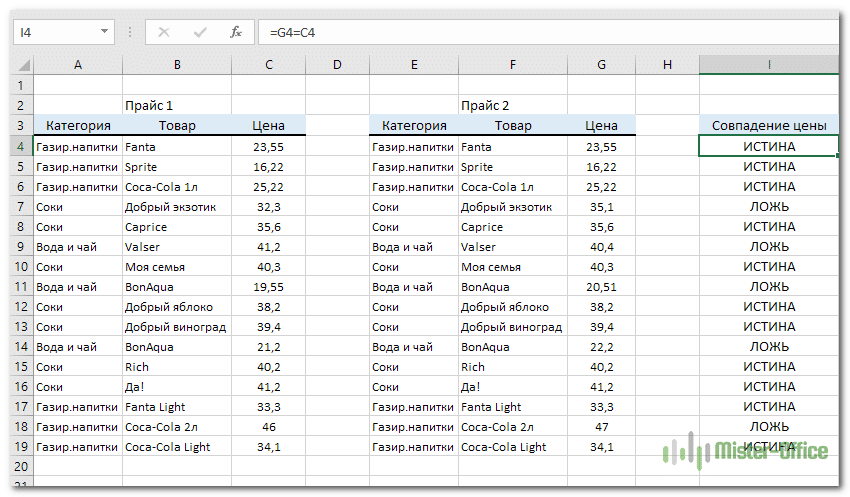 Задача элементарная, если строк в таблице немного и всего один столбец для сравнения. А сколько времени необходимо потратить если необходимо сопоставить таблицы по трем столбцам? А если строк в таблице более 1000? Надстройка поможет справиться с этой задачей гораздо быстрее.
Задача элементарная, если строк в таблице немного и всего один столбец для сравнения. А сколько времени необходимо потратить если необходимо сопоставить таблицы по трем столбцам? А если строк в таблице более 1000? Надстройка поможет справиться с этой задачей гораздо быстрее.
Добавить в Excel
Описание работы программы
После установки надстройки у вас появится новая вкладка с командой вызова функции. При нажатии на команду Сравнение диапазонов появляется диалоговое окно для ввода параметров.
Данный макрос позволяет сравнить таблицы любого объема и с любым количеством столбцов. Сравнение таблиц можно проводить по одному, двум или трем столбцам одновременно.
Диалоговое окно поделено на две части: левая для первой таблицы и правая для второй.
Чтобы сравнить таблицы необходимо выполнить следующие действия:
- Указать диапазоны таблиц.
- Установить чекбокс (галку/птичку) под выбранным диапазоном таблиц в том случае если таблица включает в себя шапку (строку заголовка).

- Выбрать столбцы левой и правой таблицы, по которым будет вестись сравнение (в случае, если диапазоны таблиц не включают заголовки столбцы будут пронумерованы).
- Указать тип сравнения.
- Выбрать вариант выдачи результатов.
Тип сравнения таблиц
Программа позволяет выбрать несколько типов сравнения таблиц:
Найти строки одной таблицы, которые отсутствуют в другой таблице
При выборе данного типа сравнения программа ищет строки одной таблицы, которые отсутствуют в другой. Если вы сопоставляете таблицы по нескольким столбцам, то результатом работы будут строки, в которых имеется различие хотя бы по одному из столбцов.
Найти совпадающие строки
При выборе данного типа сравнения, программа находит строки, которые совпадают в первой и второй таблицах. Совпадающими считаются строки, в которых значения в выбранных столбцах сравнения (1, 2, 3) одной таблицы полностью совпадают со значениями столбцов второй таблицы.
Пример работы программы в данном режиме представлен справа на картинке.
Сопоставить таблицы на основе выбранной
В данном режиме сравнения, напротив каждой строки первой таблицы (выбранной в качестве основной), копируются данные совпадающей строки второй таблицы. В случае если совпадающие строки отсутствуют, строка напротив основной таблицы остается пустой.
Сравнение таблиц по четырем и более столбцам
Если вам не хватает функционала программы и необходимо сопоставить таблицы по четырем или более столбцам, то вы можете выйти из положения следующим образом:
- Создайте в таблицах по пустому столбцу.
- В новых столбцах используя формулу =СЦЕПИТЬ объедините столбцы, ко которым хотите провести сравнение.
Таким образом, вы получите 1 столбец, содержащий значения нескольких столбцов. Ну а как сопоставить один столбец вы уже знаете.
Надстройка
VBA-Excel
Надстройка для Excel содержит большой набор полезных функций, с помощью которых вы значительно сократите время и увеличите скорость работы с программой.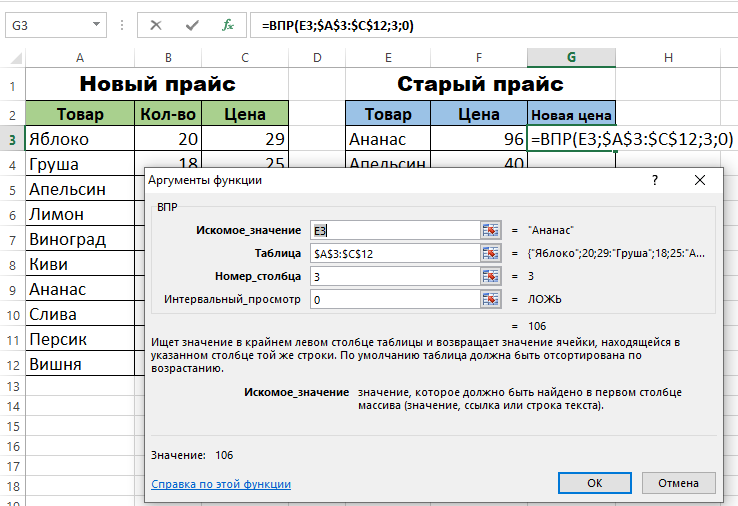
Добавить в Excel
Рекомендуем к прочтению
Удаление пустых строк и столбцов
Как вывести список уникальных значений
Формула СЦЕПИТЬДИАПАЗОН
Комментарии:
Please enable JavaScript to view the comments powered by Disqus.comments powered by Disqus
Как сравнить две таблицы с помощью OnboardingDataAssistant
В этом руководстве вы будете использовать утилиту Data AssistantA, которая задает вопросы о ваших данных, собирает информацию для описания того, что наблюдается, а затем представляет метрики и предлагает ожидания на основе ответов. для создания набора ожиданий. Коллекция проверяемых утверждений о данных. который можно использовать для определения идентичности двух таблиц. Этот рабочий процесс можно использовать, например, для проверки перенесенных данных.
Предварительные условия
- Заполнение краткого руководства.
- Работающая инсталляция «Большие надежды».
- Минимум два настроенных источника данных и активов
- Базовое понимание того, как настроить ожидание в «Больших ожиданиях»
- Завершение помощника по даннымУтилита, которая задает вопросы о ваших данных, собирает информацию для описания того, что наблюдается, а затем представляет метрики и предлагает Ожидания, основанные на ответах.
 обзор
обзор
Шаги
1. Настройка
В этом рабочем процессе мы будем использовать OnboardingDataAssistant для профилирования по запросу BatchRequestProvided к источнику данных для создания пакета. представляющие наши исходные данные, и сверить полученный набор с BatchRequest , представляющим наш второй набор данных.
Для начала нам нужно импортировать «Большие надежды» и создать экземпляр нашего контекста данных. Основная точка входа для развертывания «Большие надежды» с конфигурациями и методами для всех вспомогательных компонентов.:
импортировать большие_ожидания как gxcontext = gx.get_context()
В зависимости от вашего варианта использования, рабочего процесса и структуры каталогов вам может потребоваться обновить корневой каталог вашего контекста следующим образом:
context = gx.get_context (
context_root_dir='/my/context/root/directory/great_expectations'
)
2.
 Создание пакетных запросов
Создание пакетных запросов
Чтобы профилировать нашу первую таблицу и проверить нашу вторую таблицу, нам нужно настроить наши пакетные запросы указывая на каждый набор данных.
В этом руководстве мы будем использовать источник данных MySQL. Предоставляет стандартный API для доступа и взаимодействия с данными из самых разных исходных систем. как наши исходные данные — данные, которые мы считаем правильными.
mysql_batch_request = mysql_datasource.get_asset(
"mysql_taxi_data"
).build_batch_request()
Из этих данных мы создадим Expectation Suite — набор проверяемых утверждений о данных. и используйте этот набор для проверки нашей второй таблицы, полученной из источника данных PostgreSQL.
pg_batch_request = pg_datasource.get_asset("postgres_taxi_data").build_batch_request()
3. Исходные данные профиля
mysql_batch_request выше.
data_assistant_result = context.assistants.onboarding.run(
batch_request=pg_batch_request, exclude_column_names=["VendorID"]
)
И используйте результаты Data Assistant для создания и сохранения набора ожиданий:
expectation_suite_name = "compare_two_tables"
expectation_suite = data_assistant_result.get_expectation_suite(
expectation_suite_name=expectation_suite_name
)
context.add_or_update_expectation_suite(expectation_suite=expect ation_suite)
exclude_column_names ? Выше мы исключили "VendorID", , поэтому ожидания не будут установлены для этого столбца.
Это один из примеров того, как мы можем настроить Suite, созданный нашим Data Assistant.
Дополнительные сведения об этих конфигурациях см. в нашем руководстве по `OnboardingDataAssistant.
4. Настройка контрольной точки
Прежде чем мы сможем проверить нашу вторую таблицу, нам нужно определить контрольную точку. Основное средство проверки данных в производственном развертывании «Большие ожидания». набор ожиданий, определенный выше, до этой контрольной точки.
контрольная точка = gx.checkpoint.SimpleCheckpoint(
name="comparison_checkpoint",
data_context=context,
validations=[
{
"batch_request": pg_batch_request,
"expectation_suite_name": expectation_suite_name,
}
],
)
5.
 Val idation
Val idation
Наконец, мы можем использовать нашу контрольную точку для проверки что наши две таблицы идентичны:
checkpoint_result = checkpoint.run()
Если мы теперь проверим результаты этой контрольной точки ( results["success"] ), мы увидим, что наша проверка прошла успешно!
По умолчанию указанная выше контрольная точка также обновляет ваши документы данных, позволяя вам дополнительно проверить результаты этого рабочего процесса.
Поздравляем!
🎉 Вы только что сравнили две таблицы в источниках данных! 🎉
Систематическое сравнение активаторов транскрипции на основе CRISPR раскрывает ген-регуляторные особенности взаимодействий энхансер-промотор
1.
Джинек М., Чилински К., Фонфара И., Хауэр М., Дудна Ю.А., Шарпантье Э..
Программируемая ДНК-эндонуклеаза, управляемая двойной РНК, в адаптивном бактериальном иммунитете. Наука. 2012 г.; 337:816–821. [Бесплатная статья PMC] [PubMed] [Google Scholar]
2.
Конг Л., Ран Ф.А., Кокс Д., Лин С., Барретто Р., Хабиб Н., Хсу П.Д., У С., Цзян В., Марраффини Л.А. и др…
Мультиплексная инженерия генома с использованием систем CRISPR/Cas. Наука. 2013; 339:819–823. [Бесплатная статья PMC] [PubMed] [Google Scholar]
3.
Мали П., Ян Л., Эсвелт К.М., Аах Дж., Гуэлл М., ДиКарло Дж.Э., Норвилл Дж.Э., Черч Г.М..
Инженерия генома человека с помощью РНК с помощью Cas9. Наука. 2013; 339: 823–826. [Бесплатная статья PMC] [PubMed] [Google Scholar]
4.
Хилтон И.Б., Герсбах К.А..
Включение функциональной геномики с помощью геномной инженерии. Геном Res. 2015 г.; 25:1442–1455. [Бесплатная статья PMC] [PubMed] [Google Scholar]
5.
Мали П., Аах Дж., Стрэндж П.Б., Эсвельт К.М., Моосбернер М., Косури С., Ян Л., Черч Г.М..
Активаторы транскрипции CAS9 для скрининга специфичности мишени и парные никазы для совместной инженерии генома. Нац. Биотехнолог. 2013; 31:833–838. [Бесплатная статья PMC] [PubMed] [Google Scholar]
6.
Такоре П.И., Блэк Дж. Б., Хилтон И.Б., Герсбах К.А..
Б., Хилтон И.Б., Герсбах К.А..
Редактирование эпигенома: технологии программируемой транскрипции и эпигенетической модуляции. Нац. Методы. 2016; 13:127–137. [Бесплатная статья PMC] [PubMed] [Google Scholar]
7.
Гоэлл Дж. Х., Хилтон И. Б..
Редактирование эпигенома на основе CRISPR/Cas: достижения, приложения и клиническая полезность. Тенденции биотехнологии. 2021; 39: 678–691. [PubMed] [Google Scholar]
8.
Ци Л.С., Ларсон М.Х., Гилберт Л.А., Дудна Дж.А., Вайсман Дж.С., Аркин А.П., Лим В.А..
Перепрофилирование CRISPR в качестве управляемой РНК платформы для специфичного для последовательности контроля экспрессии генов. Клетка. 2013; 152: 1173–1183. [Бесплатная статья PMC] [PubMed] [Google Scholar]
9.
Гилберт Л.А., Ларсон М.Х., Морсут Л., Лю З., Брар Г.А., Торрес С.Е., Стерн-Гиноссар Н., Брандман О., Уайтхед Э.Х., Дудна Дж.А. и др…
CRISPR-опосредованная модульная РНК-управляемая регуляция транскрипции у эукариот. Клетка. 2013; 154:442–451. [Бесплатная статья PMC] [PubMed] [Google Scholar]
10.
Перес-Пинера П., Кочак Д.Д., Вокли С.М., Адлер А.Ф., Кабади А.М., Полштейн Л.Р., Такоре П.И., Гласс К.А., Оустераут Д.Г., Леонг К.В.и др…
Активация генов под управлением РНК факторами транскрипции на основе CRISPR-Cas9. Нац. Методы. 2013; 10:973–976. [Бесплатная статья PMC] [PubMed] [Google Scholar]
11.
Маедер М.Л., Линдер С.Дж., Касио В.М., Фу Ю., Хо К.Х., Юнг Дж.К..
CRISPR РНК-управляемая активация эндогенных генов человека. Нац. Методы. 2013; 10:977–979. [Бесплатная статья PMC] [PubMed] [Google Scholar]
12.
Таненбаум М.Э., Гилберт Л.А., Ци Л.С., Вайсман Дж.С., Вейл Р.Д..
Система мечения белков для усиления сигнала при экспрессии генов и флуоресцентной визуализации. Клетка. 2014; 159: 635–646. [Бесплатная статья PMC] [PubMed] [Google Scholar]
13.
Конерманн С., Бригам М.Д., Тревино А.Е., Юнг Дж., Абудайе О.О., Барсена С., Хсу П.Д., Хабиб Н., Гутенберг Дж.С., Нишимасу Х.и др…
Активация транскрипции в масштабе генома с помощью сконструированного комплекса CRISPR-Cas9. Природа. 2015 г.; 517: 583–588. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Природа. 2015 г.; 517: 583–588. [Бесплатная статья PMC] [PubMed] [Google Scholar]
14.
Чавес А., Шейман Дж., Вора С., Прюитт Б.В., Таттл М., Э П.Р.И., Лин С., Киани С., Гусман К.Д., Виганд Д. Джет и др…
Высокоэффективное Cas9-опосредованное программирование транскрипции. Нац. Методы. 2015 г.; 12:326–328. [Бесплатная статья PMC] [PubMed] [Google Scholar]
15.
Махата Б., Ли Дж., Кабрера А., Бреннер Д.А., Герра-Ресендес Р.С., Гоэлл Дж., Ван К., Эскобар М., Го Ю., Партасарати А.К. и др…
Компактные сконструированные модули трансактивации человека обеспечивают мощный и универсальный синтетический контроль транскрипции. 2022 г.; bioRxiv doi: 21 марта 2022 г., препринт: без рецензирования 10.1101/2022.03.21.485228. [Перекрестная ссылка]
16.
Хилтон И.Б., Д’Ипполито А.М., Вокли С.М., Такоре П.И., Кроуфорд Г.Е., Редди Т.Е., Герсбах К.А.
Редактирование эпигенома ацетилтрансферазой на основе CRISPR-Cas9 активирует гены из промоторов и энхансеров. Нац. Биотехнолог. 2015 г. ; 33: 510–517. [Бесплатная статья PMC] [PubMed] [Google Scholar]
; 33: 510–517. [Бесплатная статья PMC] [PubMed] [Google Scholar]
17.
Кернс Н.А., Фам Х., Табак Б., Генга Р.М., Сильверстайн Н.Дж., Гарбер М., Мэр Р..
Функциональная аннотация нативных энхансеров со слиянием Cas9-гистондеметилазы. Нац. Методы. 2015 г.; 12:401–403. [Бесплатная статья PMC] [PubMed] [Google Scholar]
18.
Войта А., Добринич П., Тадич В., Бокор Л., Корак П., Юлг Б., Клашич М., Золдош В..
Перепрофилирование системы CRISPR-Cas9 для целенаправленного метилирования ДНК. Нуклеиновый. Кислоты. Рез. 2016; 44:5615–5628. [Бесплатная статья PMC] [PubMed] [Google Scholar]
19.
О’Гин Х., Рен С., Николет С.М., Перес А.А., Халмай Дж., Ле В.М., Маккей Дж.П., Фарнхэм П.Дж., Сигал Д.Дж..
Редактирование эпигенома на основе dCas9 предполагает, что приобретения метилирования гистонов недостаточно для репрессии гена-мишени. Нуклеиновые Кислоты Res. 2017; 45:9901–9916. [Бесплатная статья PMC] [PubMed] [Google Scholar]
20.
Квон Д.Ю., Чжао Ю.Т., Ламоника Дж.М., Чжоу З. .
.
Локус-специфическое деацетилирование гистонов с использованием синтетического HDAC на основе CRISPR-Cas9. Нац. коммун. 2017; 8:15315. [Бесплатная статья PMC] [PubMed] [Google Scholar]
21.
Степпер П., Кунгуловски Г., Юрковска Р.З., Чандра Т., Крюгер Ф., Рейнхардт Р., Рейк В., Йелч А., Юрковски Т.П..
Эффективное направленное метилирование ДНК с помощью химерной метилтрансферазы dCas9-Dnmt3a-Dnmt3L. Нуклеиновые Кислоты Res. 2017; 45: 1703–1713. [Бесплатная статья PMC] [PubMed] [Google Scholar]
22.
Хорикоши Н., Шарма Д., Леонард Ф., Пандита Р.К., Чарака В.К., Хамбард С., Хорикоши Н.Т., Гаур Хайтан П., Чакраборти С., Кот Дж. и др…
Ранее существовавшие уровни h5K16ac в эухроматине управляют репарацией ДНК путем гомологичной рекомбинации в S-фазе. коммун биол. 2019; 2:253. [Бесплатная статья PMC] [PubMed] [Google Scholar]
23.
Ли Дж., Махата Б., Эскобар М., Гоэлл Дж., Ван К., Кхемка П., Хилтон И.Б..
Программируемое фосфорилирование гистонов человека и активация генов с использованием CRISPR/Cas9на основе хроматинкиназы. Нац. коммун. 2021; 12:896. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Нац. коммун. 2021; 12:896. [Бесплатная статья PMC] [PubMed] [Google Scholar]
24.
Залатан Дж. Г., Ли М. Э., Алмейда Р., Гилберт Л. А., Уайтхед Э. Х., Ла Русса М., Цай Дж. К., Вайсман Дж. С., Дьюбер Дж. Э., Ци Л. С. и др…
Разработка сложных программ синтетической транскрипции с использованием каркасов CRISPR РНК. Клетка. 2015 г.; 160: 339–350. [Бесплатная статья PMC] [PubMed] [Google Scholar]
25.
Чавес А., Таттл М., Прюитт Б.В., Юэн-Кэмпен Б., Чари Р., Тер-Ованесян Д., Хак С.Дж., Чекки Р.Дж., Коваль Э.Дж.К., Бухтал Дж. и др…
Сравнение Cas9активаторы нескольких видов. Нац. Методы. 2016; 13: 563–567. [Бесплатная статья PMC] [PubMed] [Google Scholar]
26.
Ченг А.В., Джиллетт Н., Ли П., Пласкон Д., Фудзивара Ю., Ван В., Тагбалут А., Ван Х..
Casilio: универсальный гибрид CRISPR-Cas9-Pumilio для регуляции генов и геномной маркировки. Сотовый рез. 2016; 26:254–257. [Бесплатная статья PMC] [PubMed] [Google Scholar]
27.
Саджван С., Маннервик М..
Активация гена с помощью dCas9-CBP и системы SAM различается по предпочтениям мишеней. науч. Респ. 2019 г.; 9:18104. [Бесплатная статья PMC] [PubMed] [Google Scholar]
науч. Респ. 2019 г.; 9:18104. [Бесплатная статья PMC] [PubMed] [Google Scholar]
28.
Так Ю.Э., Хорнг Дж.Э., Перри Н.Т., Шульц Х.Т., Айер С., Яо К., Цзоу Л.С., Арьи М.Дж., Пинелло Л., Юнг Дж.К..
Увеличение и направление CRISPR-опосредованной активации дальнего действия в клетках человека. Нац. Методы. 2021; 18:1075–1081. [Бесплатная статья PMC] [PubMed] [Google Scholar]
29.
Сюй С., Ци Л.С..
Набор инструментов CRISPR-dCas для генной инженерии и синтетической биологии. Дж. Мол. биол. 2019; 431:34–47. [PubMed] [Академия Google]
30.
Йим Ю.Ю., Тиг К.Д., Нестлер Э.Дж..
Локус-специфическое редактирование нейроэпигенома in vivo. Нац. Преподобный Нейроски. 2020; 21:471–484. [Бесплатная статья PMC] [PubMed] [Google Scholar]
31.
Шпиц Ф., Ферлонг Э.Э..
Факторы транскрипции: от связывания энхансера до контроля развития. Нац. Преподобный Жене. 2012 г.; 13:613–626. [PubMed] [Google Scholar]
32.
Бентли Д.Л.
Сочетание процессинга мРНК с транскрипцией во времени и пространстве. Нац. Преподобный Жене. 2014; 15:163–175. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Нац. Преподобный Жене. 2014; 15:163–175. [Бесплатная статья PMC] [PubMed] [Google Scholar]
33.
Кулон А., Чоу К.С., Сингер Р.Х., Ларсон Д.Р..
Эукариотическая транскрипционная динамика: от одиночных молекул к клеточным популяциям. Нац. Преподобный Жене. 2013; 14: 572–584. [Бесплатная статья PMC] [PubMed] [Google Scholar]
34.
Клемм С.Л., Шипони З., Гринлиф В.Дж..
Доступность хроматина и регуляторный эпигеном. Нац. Преподобный Жене. 2019; 20:207–220. [PubMed] [Google Scholar]
35.
Крамер П.
Организация и регуляция транскрипции генов. Природа. 2019; 573:45–54. [PubMed] [Академия Google]
36.
Джонстон С.П., Ван Н.Б., Севьер С.А., Галлоуэй К.Е..
Понимание и проектирование хроматина как динамической системы по длине и во времени. Сотовая система 2020; 11: 424–448. [PubMed] [Google Scholar]
37.
Ван Д., Гарсия-Бассетс И., Беннер С., Ли В., Су С., Чжоу Ю., Цю Дж., Лю В., Кайкконен М.У., Оги К.А. и др…
Перепрограммирование транскрипции различными классами энхансеров, функционально определяемых эРНК. Природа. 2011 г.; 474: 390–394. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Природа. 2011 г.; 474: 390–394. [Бесплатная статья PMC] [PubMed] [Google Scholar]
38.
Се С.Л., Фей Т., Чен Ю., Ли Т., Гао Ю., Ван С., Сунь Т., Суини С.Дж., Ли Г.С., Чен С. и др…
Энхансерные РНК участвуют в образовании петли, управляемой рецептором андрогена, которая избирательно усиливает активацию генов. проц. Натл. акад. науч. США 2014; 111:7319–7324. [Бесплатная статья PMC] [PubMed] [Google Scholar]
39.
Пнуэли Л., Рудницкий С., Йосефзон Ю., Меламед П..
РНК, транскрибируемая с дистального энхансера, необходима для активации хроматина на промоторе гена альфа-субъединицы гонадотропина. проц. Натл. акад. науч. США 2015; 112:4369–4374. [Бесплатная статья PMC] [PubMed] [Google Scholar]
40.
Михайличенко О., Бондаренко В., Харнетт Д., Шор И.Е., Малес М., Виалес Р.Р., Ферлонг Е.Е.М..
Степень активности энхансера или промотора отражается уровнями и направленностью транскрипции эРНК. Гены Дев. 2018; 32:42–57. [Бесплатная статья PMC] [PubMed] [Google Scholar]
41.
Ли В., Нотани Д., Розенфельд М.Г..
Энхансеры как некодирующие единицы транскрипции РНК: последние данные и перспективы на будущее. Нац. Преподобный Жене. 2016; 17: 207–223. [PubMed] [Академия Google]
42.
Карулло Н.В.Н., Филлипс III Р.А., Саймон Р.К., Сото С.А.Р., Хиндс Дж.Э., Солсбери А.Дж., Реванна Дж.С., Баннер К.Д., Янов Л., Султан Ф.А. и др…
Энхансерные РНК предсказывают регуляторные связи энхансер-ген и имеют решающее значение для функции энхансера в нейронных системах. Нуклеиновые Кислоты Res. 2020; 48:9550–9570. [Бесплатная статья PMC] [PubMed] [Google Scholar]
43.
Крейгтон М.П., Ченг А.В., Велстед Г.Г., Куистра Т., Кэри Б.В., Стайн Э.Дж., Ханна Дж., Лодато М.А., Фрэмптон Г.М., Шарп П.А. и др…
Гистон h4K27ac отделяет активные энхансеры от готовых и предсказывает состояние развития. проц. Натл. акад. науч. США, 2010 г.; 107:21931–21936. [Бесплатная статья PMC] [PubMed] [Google Scholar]
44.
Смит Э., Шилатифард А..
Биология энхансеров и энхансеропатии. Нац. Структура Мол. биол. 2014; 21:210–219. [PubMed] [Google Scholar]
Нац. Структура Мол. биол. 2014; 21:210–219. [PubMed] [Google Scholar]
45.
Прадипа М.М.
Причинная роль ацетилирования гистонов в функции энхансера. Транскрипция. 2017; 8:40–47. [Бесплатная статья PMC] [PubMed] [Google Scholar]
46.
Ян Л., Лин С., Джин С., Ян Дж. К., Танаса Б., Ли В., Меркурьев Д., Оги К. А., Мэн Д., Чжан Дж. и др…
днРНК-зависимые механизмы программ активации генов, регулируемых рецептором андрогенов. Природа. 2013; 500:598–602. [Бесплатная статья PMC] [PubMed] [Google Scholar]
47.
Фраттини В., Трифонов В., Чан Дж.М., Кастано А., Лиа М., Абате Ф., Кейр С.Т., Джи А.Х., Зопполи П., Ниола Ф.и др…
Комплексный ландшафт драйверных геномных изменений при глиобластоме. Нац. Жене. 2013; 45:1141–1149. [Бесплатная статья PMC] [PubMed] [Google Scholar]
48.
Фитц Дж., Нойманн Т., Штайнингер М., Видеманн Э.М., Гарсия А.С., Атанасиадис А., Шоберл У.Е., Паври Р..
Spt5-опосредованная транскрипция энхансера напрямую связывает активацию энхансера с физическим взаимодействием с промотором. Нац. Жене. 2020; 52: 505–515. [PubMed] [Академия Google]
Нац. Жене. 2020; 52: 505–515. [PubMed] [Академия Google]
49.
Шенфельдер С., Фрейзер П..
Контакты энхансер-промотор дальнего действия в контроле экспрессии генов. Нац. Преподобный Жене. 2019; 20:437–455. [PubMed] [Google Scholar]
50.
Туан Д., Конг С., Ху К..
Транскрипция энхансера гиперчувствительного сайта HS2 в эритроидных клетках. проц. Натл. акад. науч. США 1992 г.; 89:11219–11223. [Бесплатная статья PMC] [PubMed] [Google Scholar]
51.
Петерсон К.Р., Клегг Ч.Х., Навас П.А., Нортон Э.Дж., Кимбро Т.Г., Стаматояннопулос Г..
Влияние делеции 5’HS3 или 5’HS2 контрольной области локуса бета-глобина человека на регуляцию развития экспрессии гена глобина у трансгенных мышей с искусственной хромосомой дрожжей локуса бета-глобина. проц. Натл. акад. науч. США 1996; 93:6605–6609. [Бесплатная статья PMC] [PubMed] [Google Scholar]
52.
Эш Х.Л., Монкс Дж., Вейгерде М., Фрейзер П., Праудфут Н.Дж..
Межгенная транскрипция и трансиндукция локуса бета-глобина человека. Гены Дев. 1997 год; 11:2494–2509. [Бесплатная статья PMC] [PubMed] [Google Scholar]
1997 год; 11:2494–2509. [Бесплатная статья PMC] [PubMed] [Google Scholar]
53.
Хардисон Р., Слайтом Дж.Л., Гумуцио Д.Л., Гудман М., Стоянович Н., Миллер В..
Области контроля локуса кластеров генов β-глобина млекопитающих: объединение филогенетических анализов и экспериментальных результатов для получения функциональной информации. Ген. 1997; 205:73–94. [PubMed] [Google Scholar]
54.
Картер Д., Чакалова Л., Осборн К.С., Дай Ю.Ф., Фрейзер П..
Регуляторные взаимодействия хроматина дальнего действия in vivo. Нац. Жене. 2002 г.; 32:623–626. [PubMed] [Google Scholar]
55.
Толуис Б., Палстра Р.Дж., Сплинтер Э., Гросвельд Ф., де Лаат В..
Зацикливание и взаимодействие между гиперчувствительными сайтами в активном бета-глобиновом локусе. Мол. Клетка. 2002 г.; 10: 1453–1465. [PubMed] [Google Scholar]
56.
Денг В., Рупон Дж.В., Кривега И., Бреда Л., Мотта И., Ян К.С., Рейк А., Грегори П.Д., Ривелла С., Дин А. и др…
Реактивация генов глобина, заглушенных в процессе развития, путем принудительного образования петель хроматина. Клетка. 2014; 158:849–860. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Клетка. 2014; 158:849–860. [Бесплатная статья PMC] [PubMed] [Google Scholar]
57.
Лангмид Б., Зальцберг С.Л..
Быстрое выравнивание с промежутками чтения с бабочкой 2. Nat. Методы. 2012 г.; 9: 357–359. [Бесплатная статья PMC] [PubMed] [Google Scholar]
58.
Куинлан А.Р., Холл И.М..
BEDTools: гибкий набор утилит для сравнения геномных признаков. Биоинформатика. 2010 г.; 26:841–842. [Бесплатная статья PMC] [PubMed] [Google Scholar]
59.
Гемберлинг М.П., Сикленка К., Родригес Э., Тонн-Айзингер К.Р., Баррера А., Лю Ф., Кантор А., Ли Л., Сиглиола В., Хазлетт М.Ф. и др…
Трансгенные мыши для редактирования эпигенома in vivo с помощью систем на основе CRISPR. Нац. Методы. 2021; 18:965–974. [Бесплатная статья PMC] [PubMed] [Google Scholar]
60.
Меерс М.П., Тененбаум Д., Хеникофф С..
Вызов пика с помощью анализа разреженного обогащения для профилирования хроматина CUT&RUN. Эпигенетика Хроматин. 2019; 12:42. [Бесплатная статья PMC] [PubMed] [Google Scholar]
61.
Неф С., Куэн М.С., Рейнольдс А.П., Хауген Э., Турман Р.Э., Джонсон А.К., Райнс Э., Маурано М.Т., Виерстра Дж., Томас С. и др…
BEDOPS: высокопроизводительные операции с геномными признаками. Биоинформатика. 2012 г.; 28:1919–1920. [Бесплатная статья PMC] [PubMed] [Google Scholar]
62.
Лав М.И., Хубер В., Андерс С..
Модерированная оценка изменения кратности и дисперсии для данных секвенирования РНК с помощью DESeq2. Геном биол. 2014; 15:550. [Бесплатная статья PMC] [PubMed] [Google Scholar]
63.
Хагеге Х., Клоус П., Браем К., Сплинтер Э., Деккер Дж., Катала Г., де Лаат В., Форн Т..
Количественный анализ анализов захвата конформации хромосом (3C-qPCR). Нац. протокол 2007 г.; 2: 1722–1733. [PubMed] [Академия Google]
64.
Георгакопулос Т., Тиреос Г..
Два различных активатора транскрипции дрожжей нуждаются в функции белка GCN5 для обеспечения нормальных уровней транскрипции. EMBO Дж. 1992; 11:4145–4152. [Бесплатная статья PMC] [PubMed] [Google Scholar]
65.
Кандау Р.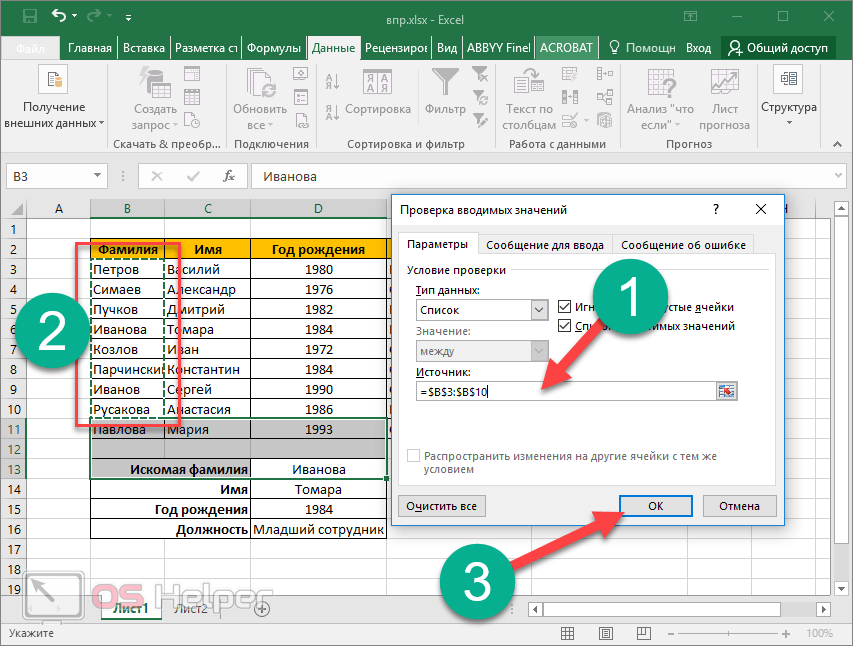 , Мур П.А., Ван Л., Барлев Н., Ин С.Ю., Розен К.А., Бергер С.Л..
, Мур П.А., Ван Л., Барлев Н., Ин С.Ю., Розен К.А., Бергер С.Л..
Идентификация белков человека, функционально консервативных с предполагаемыми адаптерами дрожжей ADA2 и GCN5. Мол. Клетка. биол. 1996 год; 16: 593–602. [Бесплатная статья PMC] [PubMed] [Google Scholar]
66.
Браунелл Дж. Э., Эллис К. Д..
Специальные HAT для особых случаев: связь ацетилирования гистонов со сборкой хроматина и активацией генов. Курс. мнение Жене. Дев. 1996 год; 6: 176–184. [PubMed] [Google Scholar]
67.
Го Дж., Ма Д., Хуан Р., Мин Дж., Е М., Ки К., Се З., На Дж..
Индуцибельная система CRISPR-ON для контролируемой активации генов в плюрипотентных стволовых клетках человека. Белковая клетка. 2017; 8: 379–393. [Бесплатная статья PMC] [PubMed] [Google Scholar]
68.
Папикян А., Лю В., Гальего-Бартоломе Дж., Якобсен С.Э..
Сайт-специфические манипуляции с локусами арабидопсиса с использованием CRISPR-Cas9системы сантегов. Нац. коммун. 2019; 10:729. [Бесплатная статья PMC] [PubMed] [Google Scholar]
69.
Ли К., Лю Ю., Цао Х., Чжан Ю., Гу З., Лю С., Ю А., Кафле П., Дикерсон К.Э., Ни М. и др…
Исследование функции энхансера путем эпигенетического редактирования CRISPR, нацеленного на энхансер. Нац. коммун. 2020; 11:485. [Бесплатная статья PMC] [PubMed] [Google Scholar]
70.
Улен М., Фагерберг Л., Халльстром Б.М., Линдског К., Оксволд П., Мардиноглу А., Сивертссон А., Кампф К., Шостедт Э., Асплунд А. и др…
Протеомика. Тканевая карта протеома человека. Наука. 2015 г.; 347:1260419. [PubMed] [Google Scholar]
71.
Чен Дж. К., Лав К. М., Голдхамер Д. Дж..
Два восходящих энхансера сотрудничают, чтобы регулировать пространственный паттерн и время транскрипции MyoD во время развития мыши. Дев. Дин. 2001 г.; 221: 274–288. [PubMed] [Google Scholar]
72.
Нордхофф В., Хюбнер К., Бауэр А., Орлова И., Малапеца А., Шолер Х.Р..
Сравнительный анализ вышерасположенных промоторов oct-4 человека, быка и мыши. Мамм. Геном. 2001 г.; 12: 309–317. [PubMed] [Академия Google]
73.
Андерссон Р., Санделин А..
Детерминанты энхансерной и промоторной активности регуляторных элементов. Нац. Преподобный Жене. 2020; 21:71–87. [PubMed] [Google Scholar]
74.
Конг С., Бол Д., Ли К., Туан Д.
Транскрипция энхансера HS2 в направлении цис-связанного гена не зависит от ориентации, положения и расстояния энхансера относительно гена. Мол. Клетка. биол. 1997 год; 17:3955–3965. [Бесплатная статья PMC] [PubMed] [Google Scholar]
75.
Ван З., Занг С., Розенфельд Дж. А., Шонес Д. Э., Барски А., Куддапах С., Цуй К., Рох Т. Ю., Пэн В., Чжан М. К. и др…
Комбинаторные закономерности ацетилирования и метилирования гистонов в геноме человека. Нац. Жене. 2008 г.; 40:897–903. [Бесплатная статья PMC] [PubMed] [Google Scholar]
76.
Консорциум проектов Encode
Интегрированная энциклопедия элементов ДНК в геноме человека. Природа. 2012 г.; 489: 57–74. [Бесплатная статья PMC] [PubMed] [Google Scholar]
77.
Дорожная карта Эпигеномика К., Кундадже А., Меулеман В., Эрнст Дж. , Биленки М., Йен А., Херави-Муссави А., Херадпур П., Чжан З., Ван Дж. и др…
, Биленки М., Йен А., Херави-Муссави А., Херадпур П., Чжан З., Ван Дж. и др…
Интегративный анализ 111 эталонных эпигеномов человека. Природа. 2015 г.; 518:317–330. [Бесплатная статья PMC] [PubMed] [Google Scholar]
78.
Чжу С., Лин Дж., Чжан Л., Пи В., Ву М., Туан Д.
Облегченный механизм отслеживания и транскрипции функции энхансера дальнего действия. Нуклеиновые Кислоты Res. 2007 г.; 35:5532–5544. [Бесплатная статья PMC] [PubMed] [Google Scholar]
79.
Мюллер Х.П., Сого Дж.М., Шаффнер В..
Энхансер стимулирует транскрипцию при присоединении к промотору через белковый мостик. Клетка. 1989 год; 58:767–777. [PubMed] [Google Scholar]
80.
Пташне М.
Генная регуляция белками, действующими вблизи и на расстоянии. Природа. 1986; 322: 697–701. [PubMed] [Google Scholar]
81.
Дэн В., Ли Дж., Ван Х., Миллер Дж., Рейк А., Грегори П.Д., Дин А., Блобель Г.А..
Контроль дальних геномных взаимодействий в нативном локусе путем целенаправленного связывания петлевого фактора. Клетка. 2012 г.; 149: 1233–1244. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Клетка. 2012 г.; 149: 1233–1244. [Бесплатная статья PMC] [PubMed] [Google Scholar]
82.
Цай П.Ф., Делл’Орсо С., Родригес Дж., Виванко К.О., Ко К.Д., Цзян К., Хуан А.Х., Саршад А.А., Виан Л., Тран М. и др…
Специфическая для мышц энхансерная РНК опосредует рекрутирование когезина и регулирует транскрипцию в транс. Мол. Клетка. 2018; 71:129–141. [Бесплатная статья PMC] [PubMed] [Google Scholar]
83.
Лай Ф., Ором Ю.А., Чесарони М., Берингер М., Таатжес Д.Дж., Блобель Г.А., Шихаттар Р..
Активирующие РНК связываются с медиатором для улучшения архитектуры хроматина и транскрипции. Природа. 2013; 494: 497–501. [Бесплатная статья PMC] [PubMed] [Google Scholar]
84.
Салдана-Мейер Р., Родригес-Эрнаес Дж., Эскобар Т., Нишана М., Хакоме-Лопес К., Нора Э.П., Брюно Б.Г., Циригос А., Фурлан-Магарил М., Скок Дж. и др…
Взаимодействия РНК необходимы для CTCF-опосредованной организации генома. Мол. Клетка. 2019; 76:412–422. [Бесплатная статья PMC] [PubMed] [Google Scholar]
85.


 обзор
обзор get_expectation_suite(
get_expectation_suite( 